Biopsies liquides et lymphomes
Le comité éditorial a sollicité des experts de biopsies liquides dans les lymphomes et précise que les techniques présentées sont en cours de développement et de validation, ne sont pas accessibles en pratique courante pour la prise en charge individuelle des patients.
La « médecine de précision » exige une connaissance détaillée du profil moléculaire du lymphome d’un patient.
La biopsie tissulaire a ses limites :
- Faisabilité et risques liés à la procédure pour les biopsies invasives thoraciques et abdominales.
- Les résultats obtenus n’intègrent pas la diversité qui existe au sein d’une tumeur ou entre 2 tumeurs d’un même patient.
Les « biopsies liquides »:
- Permettent de détecter et de caractériser les tumeurs par le biais d’une prise de sang.
- Classe émergente de méthodes permettant potentiellement d’améliorer les limites des biopsies tissulaires.
Dr Cédric Rossi
Stanford University School of Medicine, Ash Alizadeh Lab, Stanford, CA
Département d’Hématologie Hôpital Universitaire Dijon-Bourgogne
Il existe, dans le plasma, de l’ADN non contenu dans des cellules (ADN libre circulant), libéré à l’état normal par des cellules saines. Lorsqu’une tumeur est présente, elle relargue également de l’ADN qui devient accessible dans le plasma. Cet ADN tumoral circulant, ou ADNtc, ou biopsie liquide, est actuellement essentiellement analysée pour caractériser les anomalies génétiques de la masse tumorale. Cette analyse
- Résume les caractéristiques de toutes les localisations tumorales,
- Permet de détecter la présence de cellules présentant des anomalies particulièrement péjoratives (diversité intra-clonale) au diagnostic,
- Peut détecter l’émergence de clones résistants au traitement dans un prélèvement séquentiel
- Evite les risques de procédure et d’échec de biopsie des lésions résiduelles après traitement dont la nature tumorale ou non ne peut être affirmée sur les seules données d’imagerie.
Limites et pré-requis
- L’analyse de l’ADN tumoral libre, dans le plasma, les urines ou tout autre liquide environnant les tumeurs (liquide céphalo-rachidien par exemple) requiert des méthodes spécifiques, sensibles et idéalement quantitatives.
Pour être spécifique,
-
- La technique doit être capable de détecter un ensemble de mutations ciblant des gènes impliqués dans les lymphomes.
- Le biologiste doit être capable d’exclure les variants présents non pas dans la tumeur mais dans un clone anormal de l’hématopoïèse (l’hématopoïèse clonale à potentiel indéterminé (CHIP), fréquente chez le sujet âgé.
- Les analyses bioinformatiques, cruciales, permettent la suppression des erreurs liées aux techniques (par exemple, induites par la PCR).
Pour être sensible, la technique doit permettre d’identifier un nombre important de mutations
-
- Car la proportion d’ADN libre dérivé de la tumeur (ADNtc) peut être faible
- Car la détection d’au moins 1 variant caractérisé au diagnostic permet d’identifier de la maladie résiduelle après traitement.
Pour être quantitative, l’analyse doit comporter à la fois l’identification des variants, leur fréquence au sein de l’ADN libre et surtout la quantification de l’ADN libre totale.
- Cette quantité en valeur absolue d’ADN tumoral circulant reflète la quantité totale de tumeur dans l’organisme et à ce titre constitue un facteur pronostique au même titre que d’autres paramètres reflétant la masse tumorale (LDH, TMTV…)
- Les connaissances de la génétique des lymphomes, le développement de techniques performantes devraient permettre de résoudre les limites des biopsies liquides et leur transposition à grande échelle.
ADN libre = ADN tumoral circulant (dérivé de la tumeur) + ADN issu de cellules normales, + ADN issu de cellules de l’hématopoïèse clonale lorsqu’elle existe (CHIP).
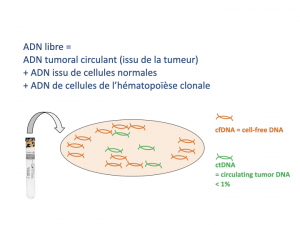
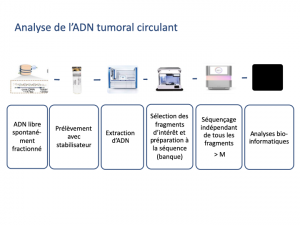
Dans l’idéal le sang doit être prélevé dans des tubes dédiés qui contiennent un stabilisateur des cellules, empêchant de « noyer » l’ADN libre circulant par l’ADN provenant des cellules du sang prélevé (plusieurs fournisseurs possibles). En l’absence de tube dédié, le sang doit être prélevé sur anticoagulant (EDTA) et le plasma doit être isolé dans les 4 heures suivant le prélèvement. L’ADN du plasma est ensuite extrait, la banque contenant l’ADN d’intérêt est préparée (voir ci-dessous) en général en 2 jours de temps de technicien, puis la banque est séquencée. Les résultats sont ensuite « moulinés » par un pipeline bioinformatique et un ensemble de variants sont proposés à l’analyse du biologiste. Ce dernier sélectionne les variants probablement présents dans la tumeur et les distingue des variants de l’hématopoïèse clonale et des polymorphismes génétiques du patient. Ces dernières tâches sont considérablement facilitées si de l’ADN non tumoral (cellules du sang si la maladie ne circule pas) est analysée en parallèle, mais compte tenu du cout de l’analyse, ceci est rarement effectué en routine.
L’ADN extrait du plasma peut être analysé par une simple PCR quantitative si une question précise est posée : détection et quantification du variant MYD88L265P par exemple dans le LCR en cas de suspicion de lymphome cérébral primitif, ou en cas de suspicion de rechute chez un patient porteur connu de la mutation [Mutter et al. J Clin Oncol 2023], ou du variant RHOA G17V dans un lymphome T type LAI [Sakata-Yanagimoto et al. Nat Genet 2014].
Mais le plus souvent des techniques de séquençage haut débit (NGS, Next Generation Sequencing) sont utilisées pour permettre l’étude de multiples locus génétiques.
Trois grandes techniques de NGS sont actuellement disponibles en routine.
- Les zones d’intérêt sont amplifiées par PCR et les produits PCR sont séquencés.
- Les zones d’intérêt sont « capturées » grâce à des sondes spécifiques, et l’ADN capturé est séquencé.
- Une troisième technique est hybride, car les zones d’intérêt sont sélectionnées par une PCR qui ne contient qu’une amorce spécifique du gène d’intérêt, l’autre amorce étant générique.
La technique 1 reste adaptée pour des petits panels (ensemble de gènes), comme ceux utilisés dans les lymphomes T. Les techniques 2 et 3 prendrons probablement rapidement une place plus importante du fait de leur plasticité (elles sont faciles à faire évoluer en fonction de l’avancée des connaissances scientifiques. Un pré-requis à une utilisation étendue en routine clinique est l’harmonisation des résultats. C’est pourquoi un consortium s’est construit autour d’experts : le projet FRENCH CONNECT, dont l’objectif à terme est de disséminer des techniques validées incluant le panel des gènes étudiés, la profondeur de séquençage requise et le pipeline d’analyse informatique.
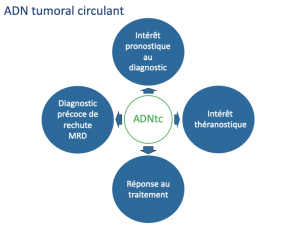
Au diagnostic, l’identification de mutations spécifiques et de sous-types moléculaires est une caractéristique clé d’une biopsie liquide ; cependant, ce n’est qu’un aspect de l’analyse de l’ADNtc dont les intérêts vont bien au-delà :
- Evaluation qualitative = génotypage (mutation, délétion, amplification/gain)
- intérêt pronostique au diagnostic en caractérisant de façon plus précise la maladie de chaque patient : classifications moléculaires [Sehn et al, N Engl J Med 2021].
- intérêt théranostique au diagnostic et/ou à la rechute en identifiant des cibles thérapeutiques potentielles,
La connaissance préalable des aberrations génétiques communes d’un lymphome spécifique permettent de définir une région cible également appelée le sélecteur. Les tests basés sur le NSG permettent d’identifier la plupart des changements génétiques somatiques qui définissent un lymphome (mutations, variants nucléotidiques simples insertions/délétions et les altérations du nombre de copies de gènes).
- Evaluation quantitative
- intérêt pronostique au diagnostic
- puis prédictif des rechutes pour les évaluations sous traitement
La capacité de l’ADNc à évaluer quantitativement la charge de la maladie a une utilité clinique potentielle importante, non seulement au moment du diagnostic, mais aussi pendant et après le traitement. En effet, la détection de la maladie résiduelle (MRD) dans le sang périphérique est devenue un outil puissant dans de nombreuses hémopathies malignes, notamment le lymphome à cellules du manteau.. Comme la plupart des lymphomes ne présentent pas de cellules lymphomateuses circulantes, peu de patients abritent des cellules tumorales circulantes (CTC) à des niveaux suffisamment élevés pour permettre la détection de la MRD à partir du sang périphérique.
Cela rend la quantification de la maladie à partir de l’ADNtc dérivé du plasma intéressante pour de nombreux sous-types de lymphomes, avec des données cliniques significatives déjà publiées pour le lymphome diffus à grandes cellules B (DLBCL) et le lymphome de Hodgkin classique (HL) en particulier.
Le génotypage des lymphomes à partir de biopsies liquides permet de classer les maladies afin d’individualiser le traitement. Dans le cas du DLBCL, cela inclut la classification issue des groupes de cellules d’origine (COO) entre les lymphomes issus des cellules B activées (ABC) et ceux issus des cellules B du centre germinal (GCB), initialement développée sur la base du profilage des gènes (GEP). D’autres méthodes de classification des COO basées sur les tissus ont été développées, en utilisant l’immunohistochimie (algorithme de Hans) ou le profilage de l’expression génétique de quelques cibles seulement avec un pouvoir discriminant élevé. La classification des COO basée sur l’identification de mutations à partir de biopsies liquides a récemment été testée dans une étude avec un taux de concordance de 80 % avec l’algorithme de Hans. Plus récemment, deux nouvelles classifications basées sur la présence ou l’absence de mutations et de variations structurelles ont été proposées par Schmitz et al. et Chapuy et al. Bien que la pertinence clinique de ces classifications ne soit pas encore établie, il est encourageant de constater qu’au moins une étude a été en mesure de classer le DLBCL dans la classification proposée par Schmitz et al. en utilisant uniquement des informations provenant de la biopsie liquide avec une concordance élevée.
De nombreuses études ont cherché à établir une « réponse moléculaire » basée sur l’ADNtc pour prédire l’évolution dans le lymphome. Comme pour les niveaux de d’ADNtc avant traitement, la plupart des données soutenant cette approche proviennent du lymphome diffus à grandes cellules B.
Chez 126 patients atteints de lymphome diffus à grandes cellules B, non traités et recevant un traitement EPOCH +/- rituximab à dose ajustée, la détection d’ADNtc par séquençage à haut débit de l’immunoglobuline (IgHTS), réalisée avant le cycle 3 du traitement a permis d’identifier un groupe de patients dont le temps de progression était inférieur [Roschewski et al. Lancet Oncol 2015].
De même, dans une étude portant sur plus de 200 sujets, Kurtz et al. ont démontré une meilleure survie sans récidive chez les patients présentant une réduction d’au moins 2 logs de l’ADNtc après un cycle de traitement par rapport aux niveaux d’avant traitement [Kurtz et al. J Clin Oncol 2018].
Il est intéressant de noter que cette réduction de 2 logs d’ADNtc après un cycle de traitement a permis d’établir un pronostic tant dans les schémas thérapeutiques de première ligne que dans les schémas de rattrapage. Cette réduction significative d’ADNtc a donc été qualifiée de réponse moléculaire précoce. De même, une réduction de 2,5 log de l’ADNtc après deux cycles de traitement s’est également avérée être de bon pronostic et a été qualifiée de réponse moléculaire majeure (RMM). Il est à noter que la RMM a une valeur pronostique plus élevée que les niveaux d’ADNc avant le traitement. En outre, la réponse moléculaire par l’ADNtc reste un facteur pronostique pour la survie sans maladie et la survie globale, même en tenant compte d’autres facteurs pertinents dans l’analyse multivariée, notamment l’IPI, la cellule d’origine et les examens TEP/CT intermédiaires. Compte tenu de l’utilité pronostique des examens TEP/CT intermédiaires dans le DLBCL, bien que leur utilité pour sélectionner le traitement des patients soit sous-optimale, la dépendance de la réponse moléculaire d’ADNtc suggère une valeur significative en tant que biomarqueur pronostique seul et donc potentiellement en combinaison avec ces examens.
Grâce à l’ensemble des technologies pouvant permettre d’analyser l’ADNtc , nous pouvons désormais envisager concrètement d’utiliser les informations qualitatives et quantitatives issues de l’ADNtc pour guider le traitement des patients (choix des drogues au diagnostic ou à la rechute) et/ou guider les stratégies de traitement selon une combinaison PET-ADNtc très pertinente pour la prise en charge des lymphomes. L’analyse de l’ADN circulant avec étude du génotype est intéressante pour le diagnostic différentiel entre lymphome médiastinal de la zone grise, lymphome à grandes cellules B primitif du médiastin et lymphome de Hodgkin classique. Pour ce dernier, l’évaluation de la réponse moléculaire au traitement via l’analyse de la maladie résiduelle (ADNtc résiduel) permet de compléter les données apportées par le TEP-scanner [Camus, Pharmaceuticals 2021].
Ce domaine d’étude est fascinant et foisonnant de résultats intéressants et il faut garder à l’esprit que le développement de ces technologies pour tirer bénéfice de l’ADNtc n’en est qu’à ses débuts. Parmi les nouveaux champs de données en cours d’exploration, on peut désormais étudier les informations immunologiques issues de l’expression des gènes [Esfahani et al. Nat Biotech 2022], ou l’ARN libre… et bien d’autres choses issues des acides nucléiques circulants dans le plasma!
Alig S, Macaulay CW, Kurtz DM, et al. Short diagnosis-to-treatment intervals is associated with higher circulating tumor DNA levels in diffuse large B-cell lymphoma. JClin Oncol 2021Aug 10;39(23):2605-2616.
Camus V, Viennot M, Lequesne J, et al. Targeted genotyping of circulating tumor DNA for classical Hodgkin lymphoma monitoring : a prospective study. Haematologica 2021 Jan 1;106(1):154-162.
Camus V, Jardin F. Cell-free DNA for the management of classical Hodgkin lymphoma. Pharmaceuticals 2021,14,207.
Chapuy B, Stewart C, Dunford AJ, et al. Molecular subtypes of diffuse large B-cell lymphoma are associated with distinct pathogenic mechanisms and outcomes. Nat Med 2018 May;24(5):679-690.
Esfahani MS; Hamilton EG, Mehrmohamadi M, et al. Inferring gene expression from cell-free DNA fragmentation profiles. Nat Biotechnol. 2022 April;40(4):585-597.
Kurtz DM, Scherer F, Jin MC, et al. Circulating tumor DNA measurements as early outcome predictors in diffuse large B-cell lymphoma. J Clin Oncol 1 oct 2018;36(28):2845-53.
Kurtz DM, Esfahani MS, Scherer F et al. Dynamic risk profiling using serial tumor biomarkers for personalized outcome prediction. Cell. 25 2019;178(3):699-713.e19.
Mutter JA, Alig SK, Esfahani MS, et al. Circulating tumor DNA profiling for detection, risk stratification, and classification of brain lymphomas. J Clin Oncol 2023 Mar 20;41(9):1684-1694.
Newman AM, Bratman SV, To J, et al. An ultrasensitive method for quantitating circulating tumor DNA with broad patient coverage. Nat Med. 2014;20(5):548-54.
Roschewski M , Dunleavy K, Pittaluga S, et al. Circulating tumour DNA and CT monitoring in patients with untreated diffuse large B-cell lymphoma: a correlative biomarker study. Lancet Oncol. 2015 May;16(5):541-9.
Scherer F, Kurtz DM, Newman AM, et al. Distinct biological subtypes and patterns of genome evolution in lymphoma revealed by circulating tumor DNA. Sci Tranl Med. 2016 Nov 9;8(364):364ra155.
Schmitz R, Wright GW, Huang DW, et al. Genetics and pathogenesis of diffuse large B-cell lymphoma. N Engl J Med 2018 April 12;378(15):1396-1407.
Sakata-Yanagimoto M, Enami T, Yoshida K , et al. Somatic RHOA mutation in angioimmunoblastic T cell lymphoma. Nat Genet 2014 Feb;46(2):171-5.
Sehn LH, Salles G. Diffuse large B-cell lymphoma. N Engl J Med. 2021 March 04;384(9):842-858.
